 >
>
Gráfico de Conceptos & Resumen usando Claude 3 Opus | Chat GPT4o | Llama 3:
Resumen:
1.- Aprendizaje de formas: Aprendizaje automático en datos que son formas, con cada punto de datos siendo una forma.
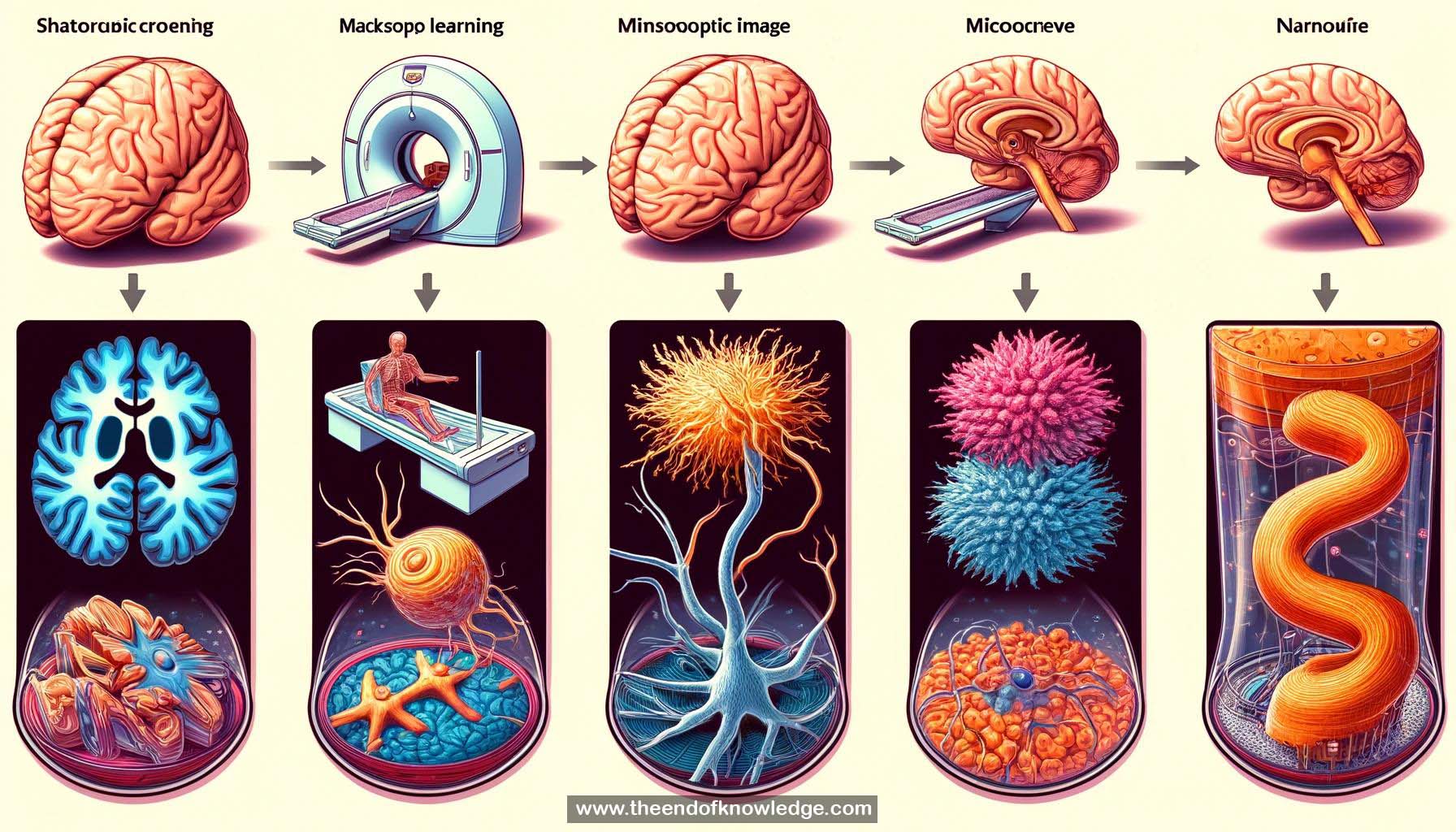
2.- Imágenes biomédicas: Estudio de estructuras biológicas a través de técnicas de imagen a varias escalas (de órganos a moléculas).
3.- Vínculo entre forma y biología: La forma de una estructura biológica está vinculada a su función y estado de salud/enfermedad.
4.- Técnicas de imagen: RMN, microscopía, crio-microscopía electrónica permiten observar formas biológicas a diferentes escalas.
5.- Preguntas biomédicas: La investigación busca responder preguntas sobre cambios de forma biológica, por ejemplo, atrofia cerebral en Alzheimer.
6.- Reconstrucción de formas: Extracción de formas de imágenes a través de algoritmos como segmentación antes del análisis.
7.- Modelado/representación de formas: Representación numérica de formas extraídas en una computadora para análisis.
8.- Variedades: Generalizaciones de espacios vectoriales que pueden ser curvadas. Muchas representaciones de formas dan lugar a variedades.
9.- Formas como variedades: Una forma en sí misma puede ser una variedad (por ejemplo, la superficie de un objeto).
10.- Espacios de formas como variedades: El espacio de todas las formas posibles de un cierto tipo forma una variedad.
11.- Espacios de transformaciones de formas como variedades: Conjuntos de movimientos o deformaciones de formas pueden ser modelados como variedades.
12.- Generalizando estadísticas a variedades: Las estadísticas tradicionales no se aplican en variedades. Las estadísticas deben ser generalizadas.
13.- Geometría de variedades como sesgo inductivo: Incorporar conocimiento de la geometría del espacio de formas puede mejorar los algoritmos de aprendizaje automático.
14.- Tipos de problemas de aprendizaje automático: Supervisado, no supervisado, aprendizaje por refuerzo, optimización - todos pueden ser generalizados a variedades.
15.- Bloques de construcción de aprendizaje en variedades: Puntos, vectores tangentes, geodésicas, distancias, mapa exponencial - usados para traducir ML a variedades.
16.- Conversión de espacio vectorial a variedades: Traducir bloques de construcción de ML permite convertir muchos algoritmos a variedades.
17.- Paquete Geomstats: Paquete de Python de código abierto que implementa cálculos en variedades para aprendizaje en variedades/geométrico.
18.- Aprendizaje en variedades de Riemann: El paquete soporta varios algoritmos en variedades de Riemann (metrizable).
19.- Variedades de posición/transformación: Ejemplo de variedad es SE(3) - el espacio de rotaciones y traslaciones 3D.
20.- Taxonomía numérica de variedades: Jerarquía/taxonomía de variedades de abstracto a concreto, implementada en geomstats.
21.- Visión general de ML adaptado a variedades: La investigación busca generalizar algoritmos de ML a través de diferentes variedades.
22.- Reducción de dimensión en variedades: Generalizando técnicas de reducción de dimensión lineal (PCA) y no lineal (autoencoders) a variedades.
23.- Vista geométrica de reducción de dimensión: Encontrar subespacios principales dentro de espacios lineales o variedades.
24.- Autoencoders variacionales (VAEs): Aprender subespacio latente no lineal del espacio de datos asumiendo modelo generativo.
25.- Generalizando VAEs a variedades: Adaptando componentes de VAE (modelo generativo, función de pérdida) a versiones de variedades.
26.- Componentes de Manifold VAE (mVAE): El mapa exponencial reemplaza la suma. La distancia geodésica reemplaza la euclidiana. Funciones de pérdida adaptadas.
27.- mVAE vs alternativas: mVAE más rápido que el método de reducción de dimensión en variedades basado en MCMC.
28.- mVAE explica la curvatura del espacio latente de VAE: La perspectiva de variedades muestra por qué los VAEs tienden a aprender espacios latentes más planos.
29.- Pipeline general de análisis de formas: Extracción de formas, modelado, cálculo en variedades, generación de ideas.
30.- Investigación en curso: Generalizar aún más ML a variedades, aplicando a datos de formas biomédicas.
Bóveda de Conocimiento construida porDavid Vivancos 2024